利用CIPRES云计算构建最大似然进化树
<1> 利用 makeblastdb 构建本地blast数据库
<2> 利用 blastall 在本地数据库中进行blast
除了使用blastall软件之外,也可以使用blast+,参照利用NCBI的Blast+进行本地化序列相似性检索。
1 | blastall -p blastn -d /path/to/database/blast.db -i query.fasta -o output.txt -e 1e-10 -m 8 -b 250 -a 20 |
blastall程序的常用参数说明:
-d 设置通过makeblastdb构建的本地数据库
-i 输入序列文件,FASTA格式
-o 程序输出结果文件
-b 程序计算保留前多少条序列,如本次为250,则表示结果保留最相似的250条序列
-e 期望值,这一参数控制搜索的灵敏度,可以输入整数(如100),分数 (如1/100),小数 (如0.001)或是指数 (如5e-5),默认值是10.0。
-m 设定搜索结果的显示格式,m参数的选项有12个
-a 程序运行调用的核数目
-m 参数备选值以及对应意义
-m 0 默认参数,显示query和subject两两比对的信息
-m 1 显示query在所有subjects上的定位信息,并显示一致性比对信息,subject之间不同的碱基/氨基酸会被标出
-m 2 显示query在所有subjects上的定位信息但是不显示一致性比对信息,subject之间不同的碱基/氨基酸会被标出
-m 3 显示query在所有subjects的定位和一致性比对信息,不显示subjects之间的差异
-m 4 显示query在所有subjects上的定位信息但是不显示一致性比对信息,不显示subjects之间的差异
-m 5 显示query在所有subjects上的定位信息但是不显示每个碱基/氨基酸的比对信息,补充“-”比对区域,subjects之间不同的碱基/氨基酸会被标出
-m 6 显示query在所有subjects上的定位信息但是不显示每个碱基/氨基酸的比对信息,补充“-”对齐比对区域,不显示subjects之间的差异
-m 7 输出XML格式的blast结果
-m 8 用列表格式显示比对结果。从左到右各列的意义依次是:query名, subject名,一致性百分数,比对长度,错配数,空位数,query比对起始位 点和终止位点,subject比对起始位点和终止位点,期望值,比对得分
-m 9 用带有注释行的列表格式显示比对结果,格式与-m 8一样,只是在每 个query的必读结果前面加了注释行用于说明列表中各列的意义
-m 10 输出文件为ASN格式的文本文件
-m 11 输出文件为二进制文件
<3> 提取blast结果序列
blastall程序在选择-m 8或-m 9参数后的结果文件中,含有blast得到的序列ID信息。这一步骤就是根据这个序列ID,提取序列并保存为FASTA格式。
提取的程序脚本和方法参照Extract sequences from fasta file by name。
<4> 多序列比对
以提取出来的序列作为输入文件,利用Cluster Omega软件进行多序列比对。EBI网页平台提供了在线Cluster Omega计算服务;也可以在本地服务器中安装Cluster Omega软件。
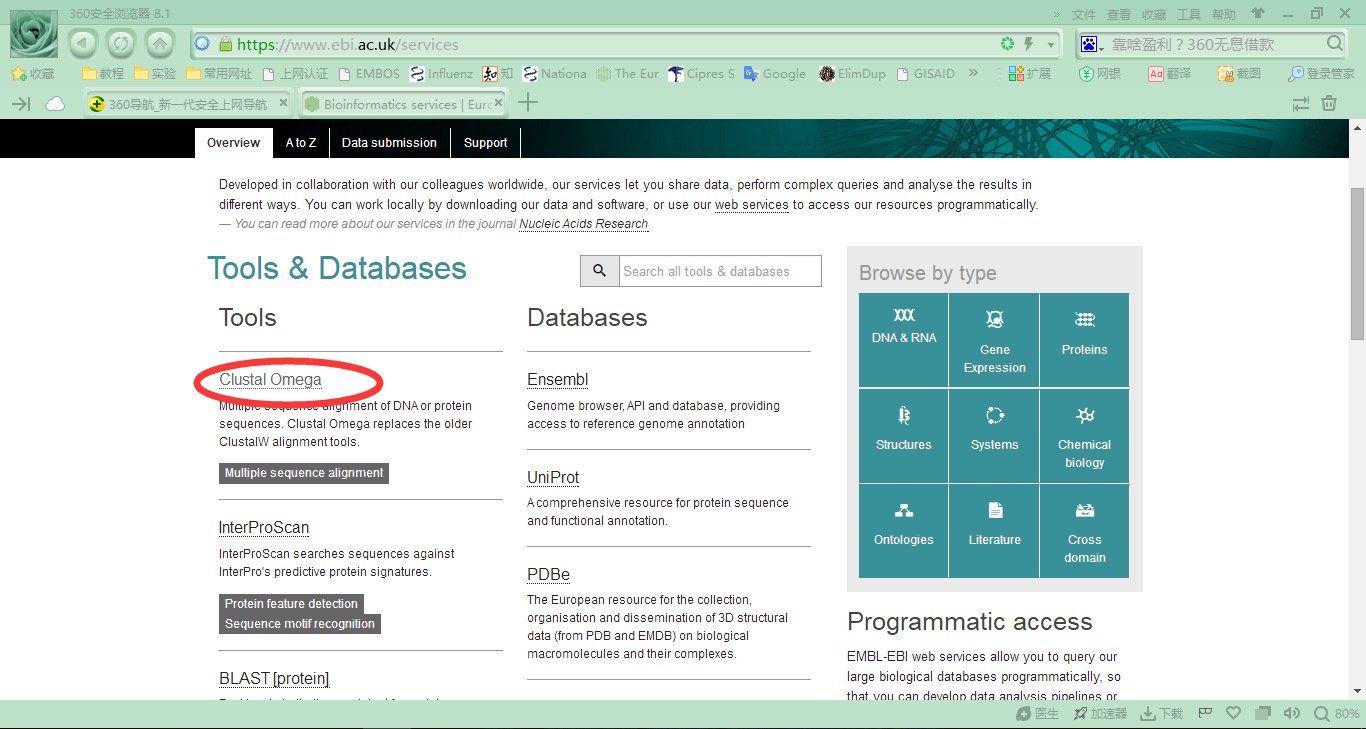
<5> 手动修剪比对后的序列文件
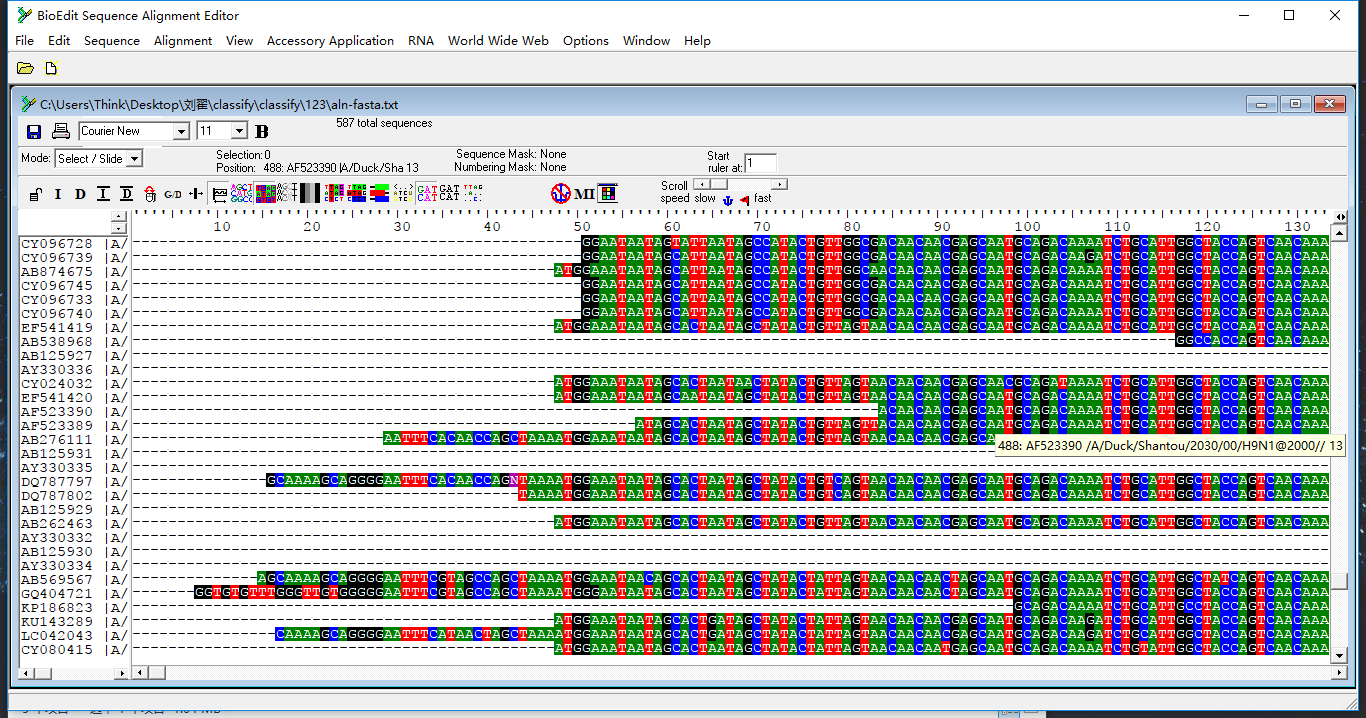
在BioEdit程序中打开比对后序列文件,如下图。
为了减少计算误差,需要删除序列两端参差不齐的部分序列。这一过程比较主观,其中一种方法是删除起始密码子(ATG)之前的序列,以及终止密码子之后的序列。
<6> 构建最大似然进化树前的准备
(A) 在构建进化树之前,需要通过JmodelTest软件计算适合该数据的最优模型。
(B) 本次使用的构建程序为 CRISPR 云计算平台,该工具对序列ID中的特殊符号敏感,即ID中不能含有逗号、空格、分号等符号。因此需要使用Notebook ++等工具替换掉。
<7> 使用CIPRES云计算平台构建最大似然进化树
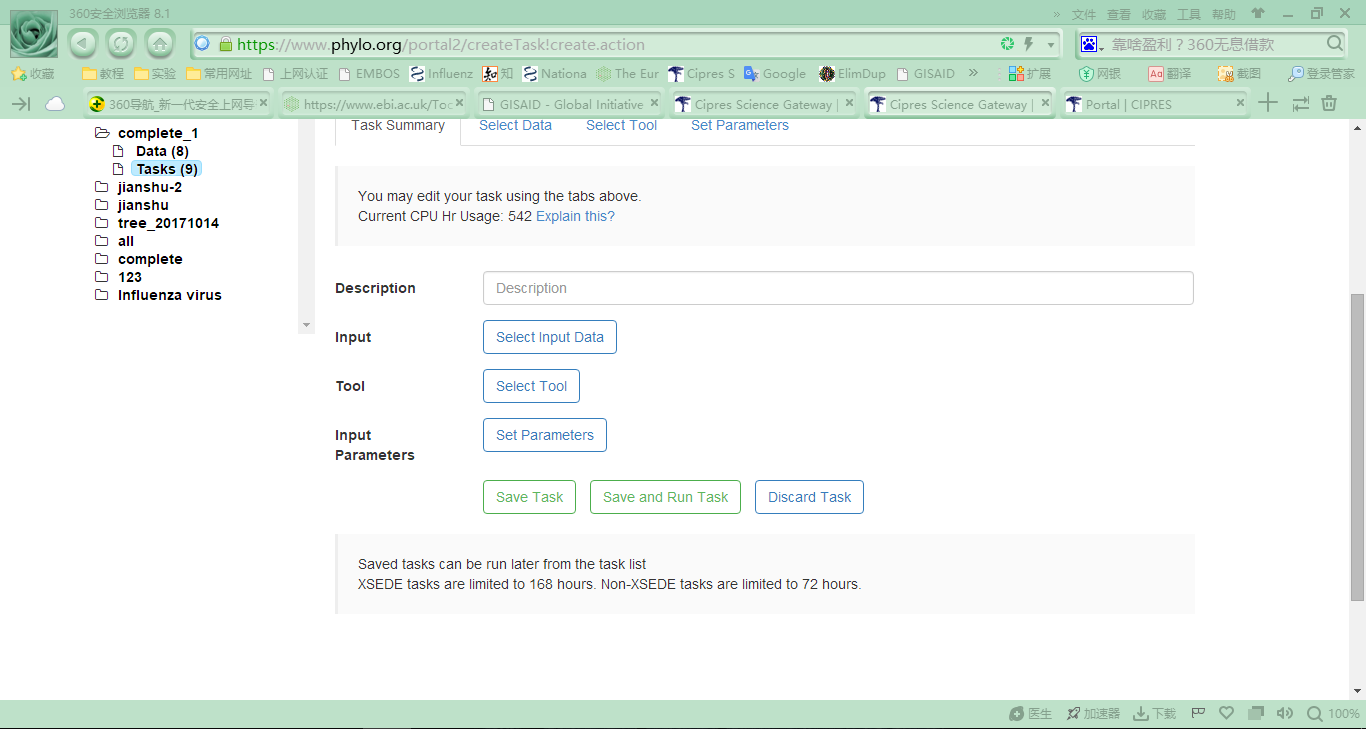
(A) 使用个人邮箱注册CIPRES云计算平台;
(B) 在云平台创建任务,并将准备好的序列文件上传至云平台。
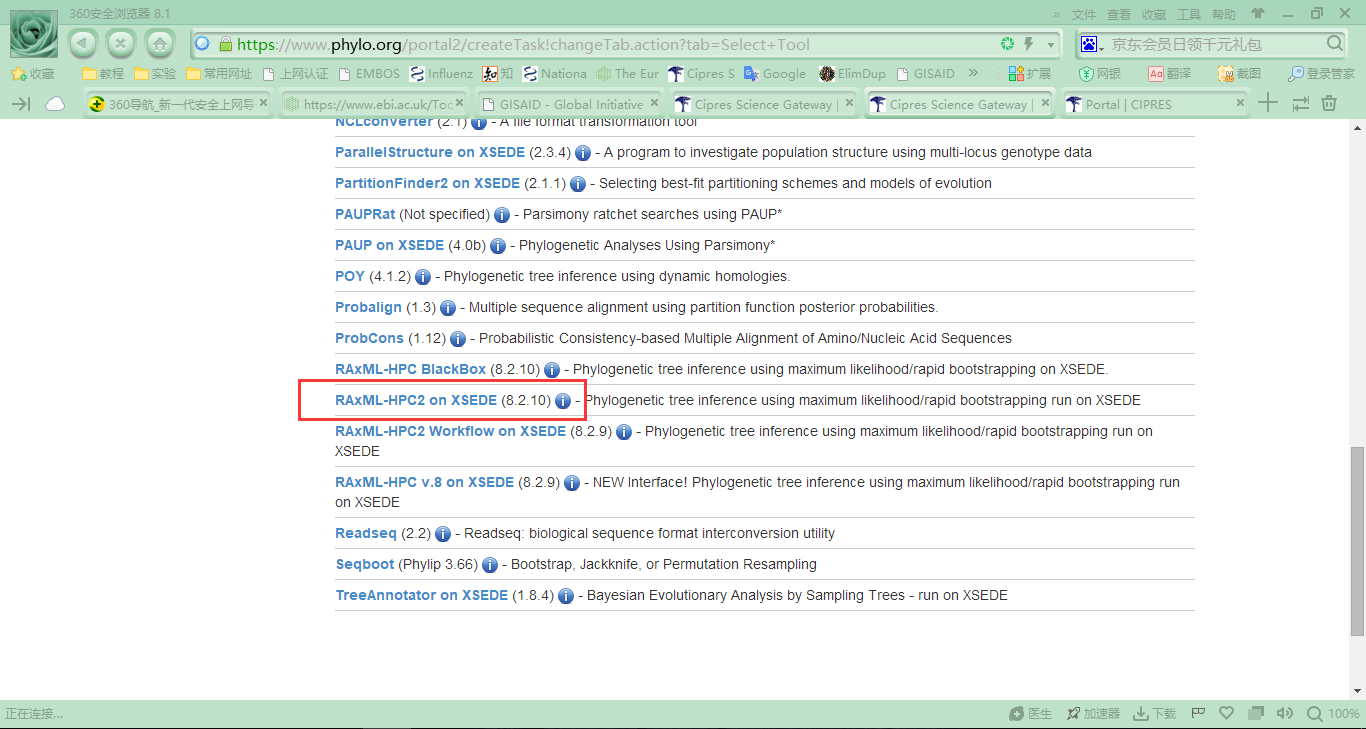
(C) 选择计算程序和设置程序参数。这里,我们选择RaxML-HPC2 on XSEDE作为程序。并在参数设置中,计算模型选择JmodelTest计算出来的最优模型;Bootstrap iteration参数设置为1000。
(D)待计算完成后,下载进化树文件。为了适用进化树可视化软件Figtree,需要对文件做格式转换。即在Notebook++等支持正则表达式的软件中,:([\.\d]+)\[(\d+)\]替换为$2:$1。
原文作者: Billy & Barney
原文链接: https://liangbilin.github.io/2020/04/20/Billy--利用CIPRES云计算构建最大似然进化树/
版权声明: 转载请注明出处(必须保留作者署名及链接)