表观组染色体开放区域分析:ATAC-seq
DNA的高维空间结构
人类细胞DNA完全展开约有2米长,那么细胞是如何将这些DNA装入直径仅为5微米大小的细胞核的呢?细胞会将DNA进行不同层次的折叠压缩形成可以调控的结构。具体而言,DNA先缠绕在组蛋白形成核小体结构;然后6个核小体形成一个环,组成螺线管;螺线管进一步折叠成超螺线管;超螺线管再缠绕形成染色体。
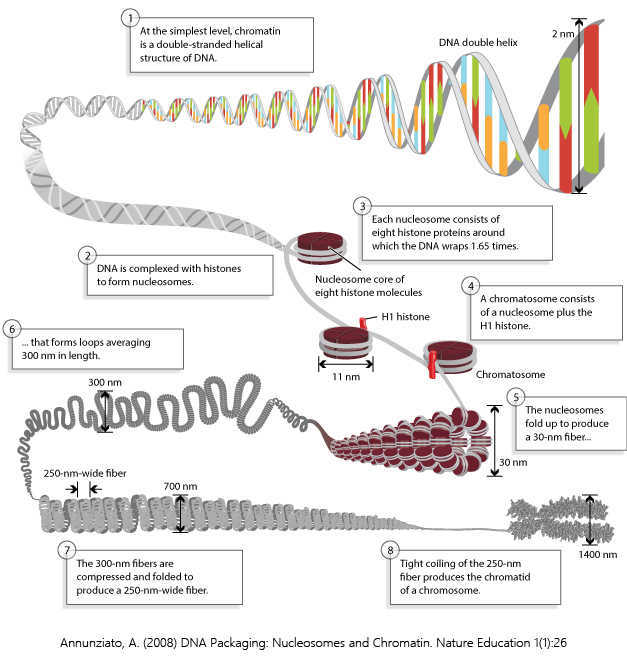
当某个基因需要表达的时候,涉及到的区域就会从压缩的高级结构解开。这部分打开的区域就叫做开放染色质(open chromatin),一些调控蛋白如转录因子或辅因子就能够与之结合,染色质的这种特性就被称为可进入性(chromatin accessibility)。
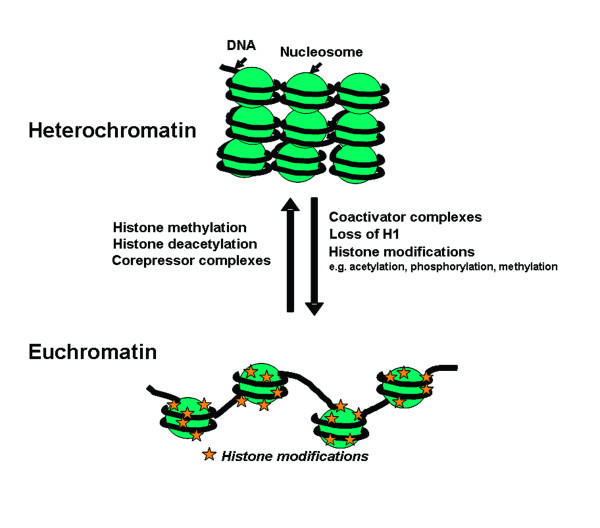
开放染色质区域鉴定方法
1. 传统实验方法:MNase-seq 和 DNase I hypersensitivity assay
这两个实验的主要思路是一致的:染色质变得开放,就意味着DNA和组蛋白的浓聚程度降低,就会有一部分DNA暴露出来。而一旦失去了蛋白质的保护,这部分DNA就可以被DNA酶(MNase或DNase I)切割。再把切割出来的DNA进行测序并比对到参考基因组,就能知道开放染色质的区域。但是这两个方法的重复性很差且耗时费力。
2. DNA转座酶结合高通量测序技术的新方法:ATAC-seq
2013年,美国Stanford大学的William Greenleaf教授研发了一种全新的方法,即 Assay for Transposase Accessible Chromatin with high-throughput sequencing (ATAC-seq)。
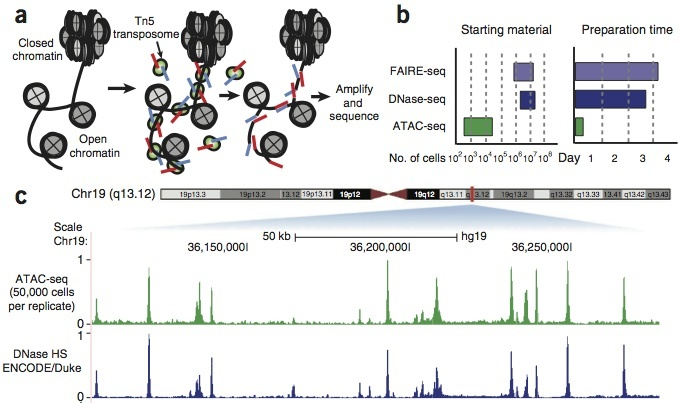
DNA转座是通过DNA转座酶实现的,把DNA序列从染色体的一个区域搬运到另外一个区域的现象。它也要求相应区域是开放的。那么,如果将携带已知DNA序列标签的转座复合物(即带着测序标签的转座酶)加入到细胞核中,序列标签将插入到基因组上。再利用标签上已知的序列进行PCR扩增和测序,就能够获得开放染色质区域的信息。相比于传统方法,ATAC-seq的重复性和可操作性都更高,而且对细胞量要求也更小,最少500多个细胞即可。
单细胞水平ATAC-seq
目前大部分研究是在群体细胞水平中探索表观遗传的平均特征,但极其复杂和富有异质性的组织则要求分辨率更高的单细胞表观遗传学研究技术。2015年, William Greenleaf 团队将 ATAC-seq 方法与 Fluidigm 的单细胞平台C1整合,利用微流控芯片完成捕获、裂解、转座、PCR等实验过程,建立了自动化的单细胞染色质可接近性图谱研究方法 Single-cell ATAC-seq (scATAC-seq)。
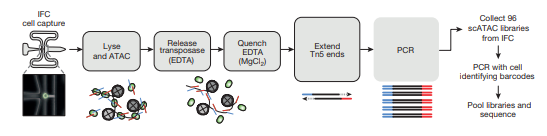
ATAC技术获得染色质开放区域信息主要依赖于Tn5转座酶,这是一种随机切割DNA的酶。其原理是核小体连接致密的地方,转座酶不能进入,而染色质开放的区域,转座酶能够进入并切割暴露的DNA,同时连接上特异性的序列。因此,只要将携带已知DNA序列标签的转座酶加入到细胞核中,再利用已知序列的标签进行PCR后测序,就可获得基因组染色质开放程度的信息。
参考资料
原文作者: Billy & Barney
原文链接: https://liangbilin.github.io/2019/12/01/Billy--表观组染色体开放区域分析—ATAC-seq/
版权声明: 转载请注明出处(必须保留作者署名及链接)