使用poretools处理fast5格式数据
目前三代测序主要为PacBio公司的SMRT测序技术和Oxford Nanopore Technology的纳米孔测序技术。相比于第一代和第二代测序技术,三代测序技术最为核心的特点就是单分子和长读长。第三代测序下机数据以fast5 (本质上为HDF5格式)格式存储。
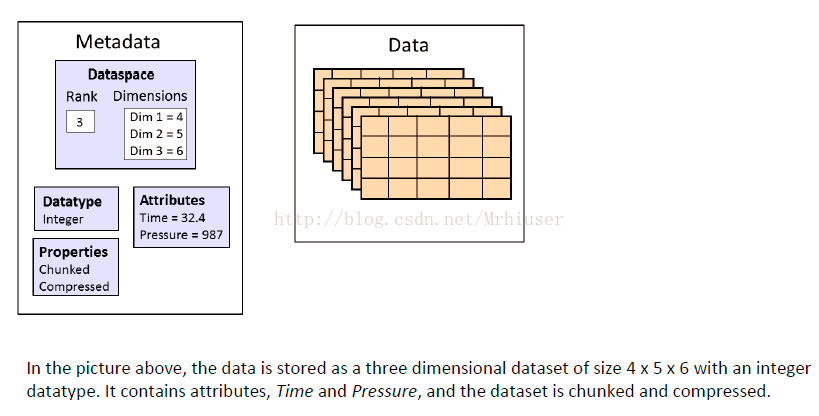
HDF5的文件组织:一个HDF5文件就是一个由两种基本数据对象(groups and datasets)存放多种科学数据的容器:
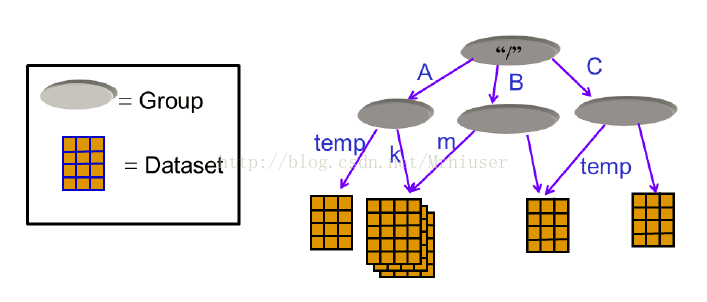
- HDF5 group:包含0个或多个HDF5对象以及支持元数据(metadata)的一个群组结构。
- HDF5 dataset:数据元素的一个多维数组以及支持元数据(metadata)。
(一) 安装anaconda(或 minianaconda)
网上很多教程,此处省略…
(二) 通过conda安装poretools
- 创建虚拟环境
1 | conda create -p ~/envs/poretools |
- 检索bioconda中收录的poretools版本信息
1 | conda search poretools |
- 根据检索结果,确定下载的版本
1 | conda install -p ~/envs/poretools poretools=0.6.0 |
- 使用之间,激活该虚拟环境
1 | conda activate ~/envs/poretools |
(三) 使用教程
- 提取FASTQ格式序列
1 | poretools fastq test.fast5 |
- 提取FASTA格式序列
1 | poretools fasta test.fast5 |
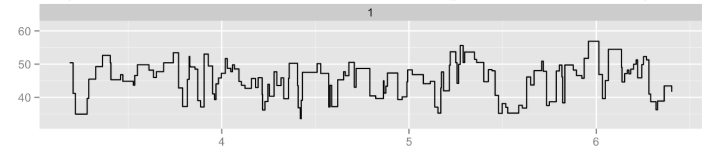
- 绘制电信号曲线
1 | poretools squiggle test.fast5 |
- 其他测序指标统计,参照官方资料
参考资料
https://poretools.readthedocs.io/en/latest/content/examples.html#poretools-fastq
原文作者: Billy & Barney
原文链接: https://liangbilin.github.io/2019/09/21/Billy--使用poretools处理fast5格式数据/
版权声明: 转载请注明出处(必须保留作者署名及链接)