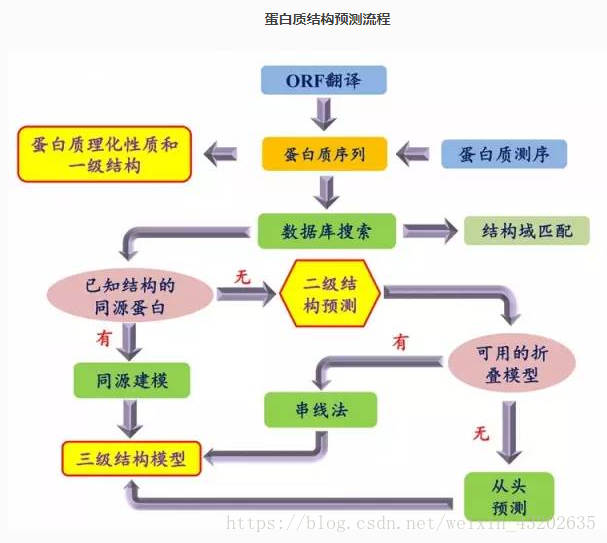
蛋白结构同源建模
无论在药物设计还是分子模拟等研究中,蛋白质结构信息都是必需的。结构生物学领域能够利用扫描电镜,核磁共振以及X衍射等手段获取大多数蛋白质的结构。然而,一方面,蛋白质结构的解析工作需要专业平台来完成,可是很多实验团队并不具备这样的平台;另一方面,在如疾病暴发等应急情景下需要迅速获取蛋白结构信息。此时,我们可以选择蛋白质结构预测的方式开展工作。
蛋白质结构预测分为同源建模和从头预测两大类。同源建模是指利用具有相似氨基酸序列且蛋白结构信息已知的蛋白质作为模板,来进行蛋白结构模型的构建。从头预测则是指不依赖于模板,仅从氨基酸序列信息进行蛋白结构模型的构建。实践表明,同源建模预测的准确度高于从头预测。可实现同源建模的工具有Swiss Model、INSIGHTii、modeller、spdbvierwer等等。
利用 Swiss Model 构建蛋白结构模型
Swiss Model 是巴塞尔大学推出的一款在线同源建模服务器,其使用非常简单方便,只需要将序列粘入,即可自动化处理结果,下面是其简单的使用方法:
第一步,打开 Swiss Model的官方网页,可以看到如下界面。点击 Start Modelling 开始进行模型构建。
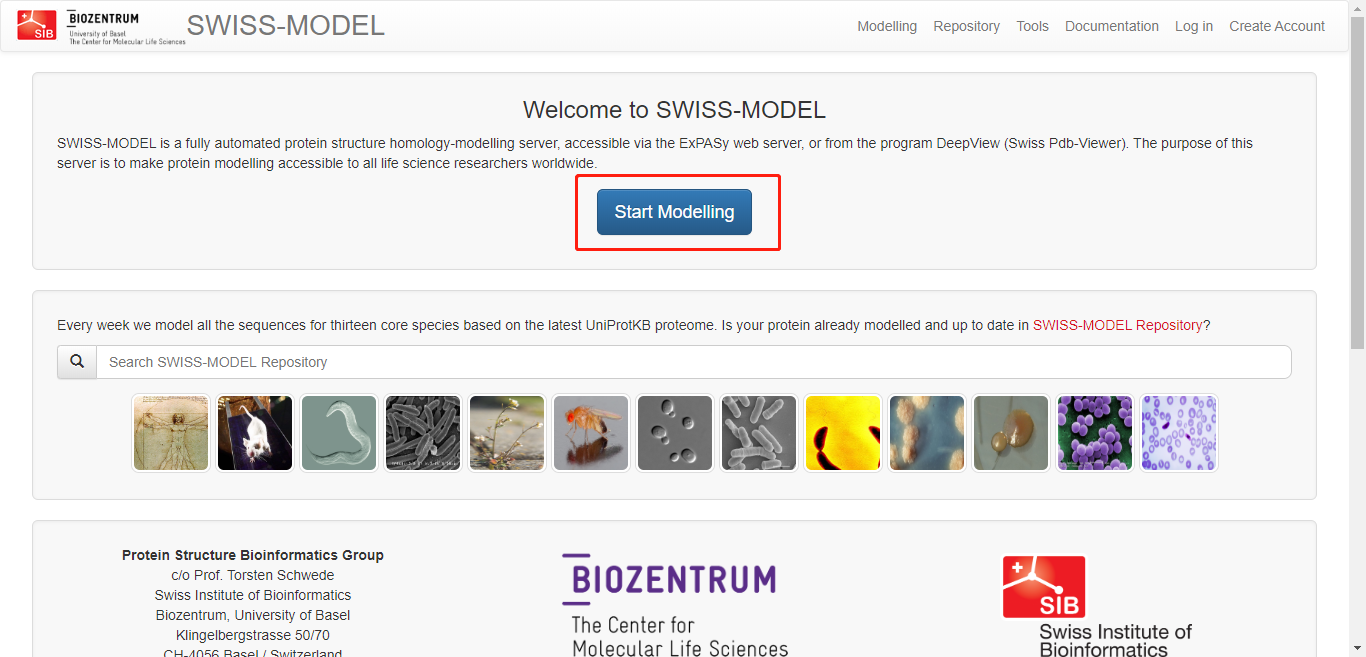
Swiss Model 的官方网页界面
第二步,在新的界面输入氨基酸序列或上传包含序列的文件。然后,点击 Search For Templates 开始检索模板信息。
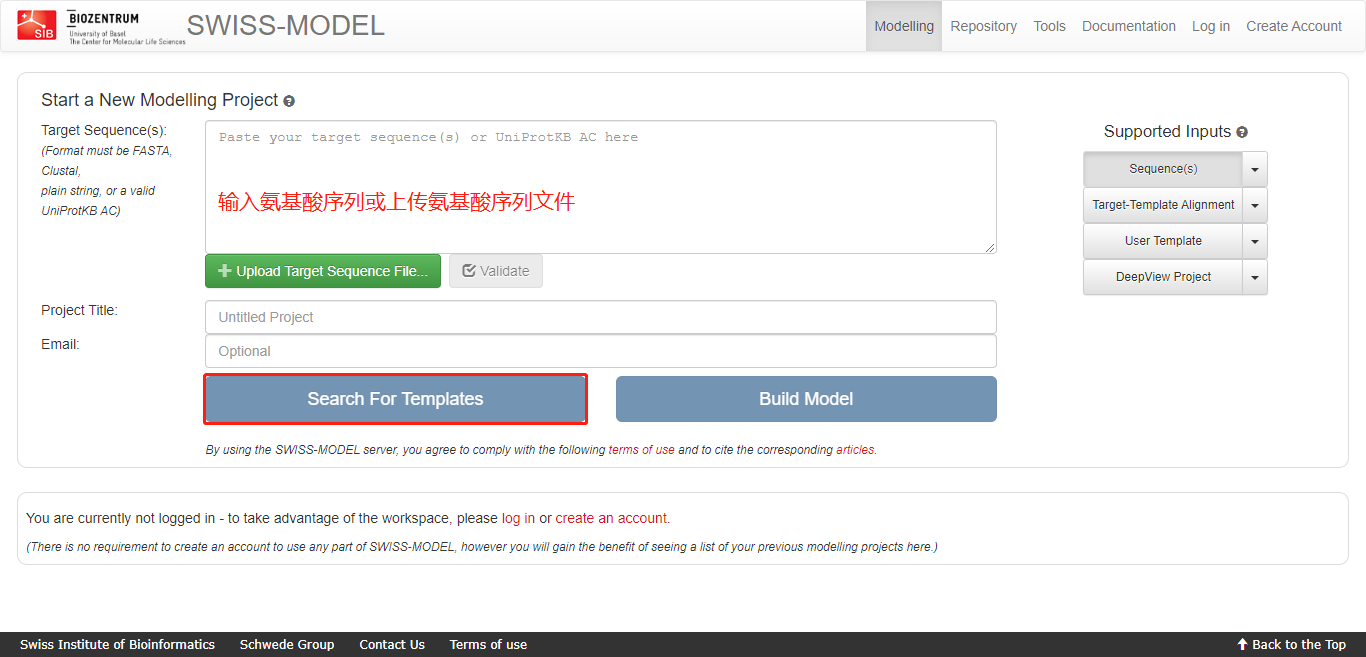
这里提供一个氨基酸测试序列:
1 | MDEMATTQISKDELDELKEAFAKVDLNSNGFICDYELHELFKEANMPLPGYKVREIIQKLMLDGDRNKDGKISFDEFVYIFQEVKSSDIAKTFRKAINRKEGICALGGTSELSSEGTQHSYSEEEKYAFVNWINKALENDPDCRHVIPMNPNTDDLFKAVGDGIVLCKMINLSVPDTIDERAINKKKLTPFIIQENLNLALNSASAIGCHVVNIGAEDLRAGKPHLVLGLLWQIIKIGLFADIELSRNEALAALLRDGETLEELMKLSPEELLLRWANFHLENSGWQKINNFSADIKDSKAYFHLLNQIAPKGQKEGEPRIDINMSGFNETDDLKRAESMLQQADKLGCRQFVTPADVVSGNPKLNLAFVANLFNKYPALTKPENQDIDWTLLEGETREERTFRNWMNSLGVNPHVNHLYADLQDALVILQLYERIKVPVDWSKVNKPPYPKLGANMKKLENCNYAVELGKHPAKFSLVGIGGQDLNDGNQTLTLALVWQLMRRYTLNVLEDLGDGQKANDDIIVNWVNRTLSEAGKSTSIQSFKDKTISSSLAVVDLIDAIQPGCINYDLVKSGNLTEDDKHNNAKYAVSMARRIGARVYALPEDLVEVKPKMVMTVFACLMGRGMKRV |
模板检索结果如下:
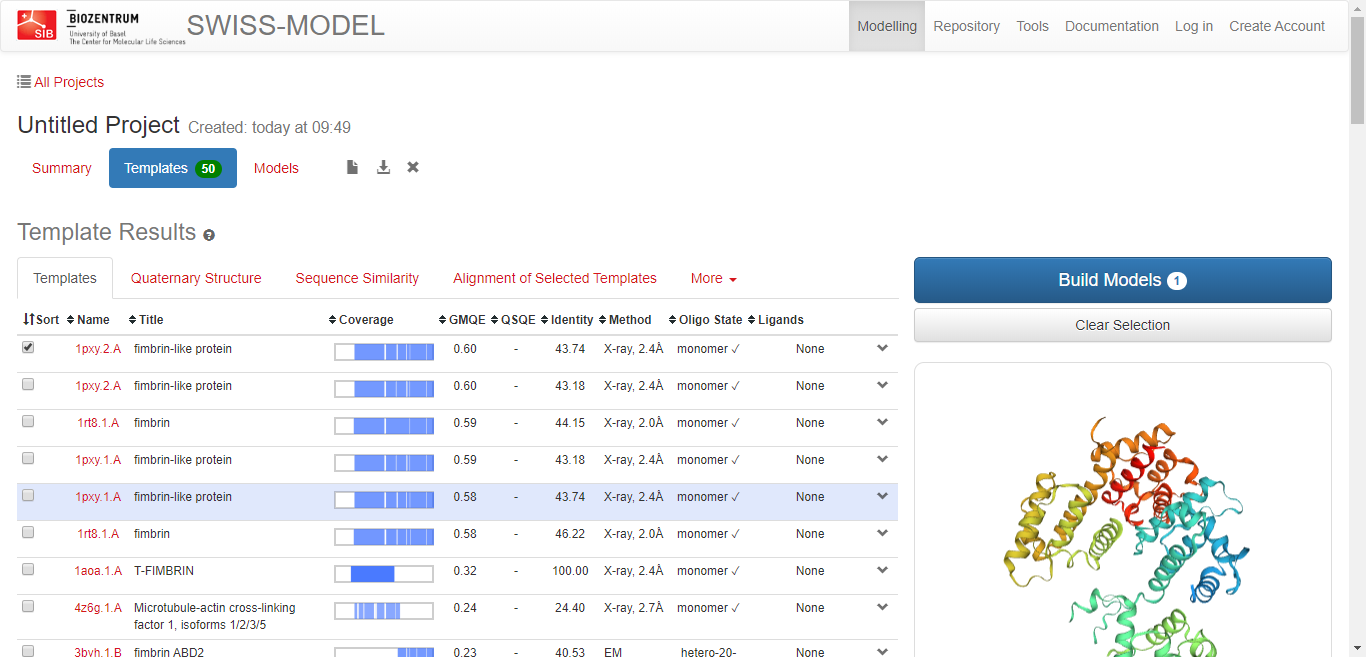
第三步,勾选模板之后,点击 Build Models 进行建模。
作为测试,只选第一个模板进行模型构建。真正实践过程中,可适当多选择几个模板进行构建,然后进行评估,以确定最佳模型。
建模结果如下:
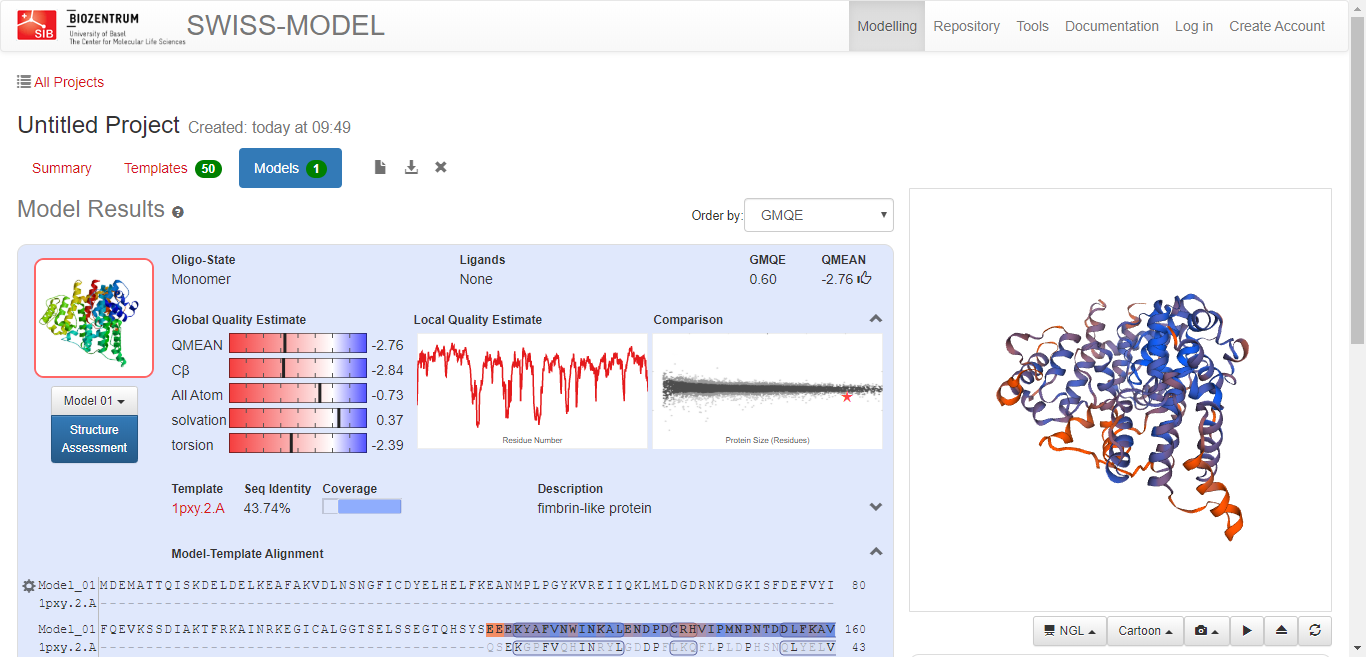
最后,可以将模型下载下来,使用 PyMOL 软件进行可视化。
原文作者: Billy & Barney
原文链接: https://liangbilin.github.io/2019/07/10/Billy--蛋白结构同源建模/
版权声明: 转载请注明出处(必须保留作者署名及链接)